Nhận diện và đánh giá tính chống chịu mặn của các giống lúa mùa dựa trên dấu phân tử SSR (Simple Sequence Repeats)
Nghiên cứu do các tác giả: Trần Hữu Phúc, Vũ Anh Pháp - Viện Nghiên cứu Phát triển Đồng bằng sông Cửu Long, Trường Đại học Cần Thơ, Nguyễn Lam Minh, Trần Thị Xuân Mai - Viện Nghiên cứu và Phát triển Công nghệ Sinh học, Trường Đại học Cần Thơ và Phạm Văn Mịch - Trung tâm Giống Nông nghiệp Cà Mau thực hiện.
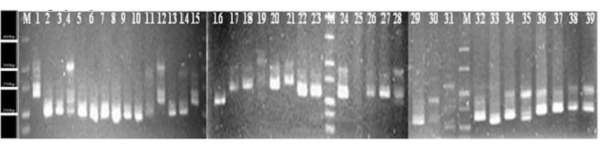
Sản phẩm PCR với dấu phân tử RM3412
Theo kết quả sưu tập nguồn giống tại đồng ruộng năm 2016, trong các tỉnh vùng Đồng bằng sông Cửu Long, Tỉnh Cà Mau có nhiều giống lúa mùa bản địa chiếm 82% tổng số mẫu giống thu thập được, nổi tiếng như: Tài Nguyên, Ba Bông Mẵn, Trắng Bồ Câu, Nàng Co Đỏ, Tép Hành, Trắng Tròn, Trắng Mây, … Các giống này được canh tác chủ yếu trong vùng nhiễm mặn với mô hình lúa tôm, lúa cá và chống chịu tốt với điều kiện mặn. Trong những năm gần đây, số lượng và diện tích trồng các giống lúa trên bị suy giảm nhanh chóng làm nguy cơ thất thoát nguồn gen di truyền quý giá. Vì vậy, đánh giá, bảo tồn và phát triển các nguồn gen lúa bản địa này rất có ý nghĩa không chỉ trong hiện tại mà còn cho tương lai.
Hiện nay, các tiến bộ về công nghệ sinh học đã được ứng dụng rộng rãi trong công tác chọn tạo giống, trong đó dấu phân tử các chuỗi lặp lại đơn (simple sequence repeats- SSR) đã được sử dụng phổ biến do đặc tính phong phú, phân phối rải đều trên toàn bộ gen lúa, rất đa hình so với các dấu phân tử khác và hơn hết, SSR còn có tính chuyên biệt cao (Miah et al., 2013). Do đó, SSR đã trở thành dấu phân tử di truyền quan trọng trong công tác chọn giống lúa. Mặc dù các dấu SSR liên kết với tính chống chịu mặn được tìm thấy trên tất cả 12 cặp nhiễm sắc thể của lúa, sự phân bố của chúng không đồng đều, tần số hiện diện cao nhất của các dấu SSR nằm trên nhiễm sắc thể số 1 và ít nhất là nhiễm sắc thể thứ 10 (Molla et al., 2015). Mục tiêu của nghiên cứu này nhằm nhận diện tính chịu mặn của các giống lúa mùa, dựa trên kiểu hình, từ đó tìm ra dấu SSR liên kết chặt với kiểu gen chống chịu mặn Saltol trên nhiễm sắc thể số 1.
Thí nghiệm được thực hiện trên 38 giống lúa, trong đó 35 giống lúa mùa trồng ở tỉnh Cà Mau, 01 giống ở Sóc Trăng, giống IR28 được dùng làm đối chứng (chuẩn nhiễm mặn) và giống Pokkali được dùng làm đối chứng (chuẩn chịu mặn). Các giống lúa mùa được cung cấp bởi Viện Nghiên cứu Phát triển Đồng bằng sông Cửu Long, Trường Đại học Cần Thơ. Để nhận diện dấu phân tử liên kết chặt với gen chịu mặn các giống lúa mùa, cả kiểu hình và kiểu gen chịu mặn giai đoạn mạ, nhóm nghiên cứu đã thanh lọc tính chịu mặn ở mức EC = 12 mS/cm theo quy trình của Viện Nghiên cứu Lúa quốc tế. Kết quả thí nghiệm có 23,7% giống được đánh giá là chịu mặn, 28,9% chịu mặn trung bình, 39,5% nhiễm mặn và 7,9% rất nhiễm mặn. Về kiểu gen, 2 dấu phân tử các chuỗi lặp lại đơn (simple sequence repeats-SSR) là RM493 và RM3412được chọn để nhận diện kiểu gen chịu mặn. Hiệu quả đánh giá bằng RM493 và RM3412 đạt từ 70% trở lên ở tất cả các nhóm cấp độ chịu mặn. Dựa trên hai dấu SSR này, 36 giống lúa mùa được chia thành 7 nhóm chính theo phương pháp UPGMA. Nhóm I có các giống có khả năng chịu mặn khá. Các giống có kiểu gen chịu mặn trung bình hầu hết thuộc nhóm II và III. Các kiểu gen nhiễm và rất nhiễm mặn thuộc các nhóm từ IV đến VII. Kết quả RM493 và RM3412 là dấu phân tử hữu ích trong chọn lọc các giống lúa mùa chịu mặn.
Tạp chí Khoa học Trường ĐH Cần Thơ-Tập 54, Số 6-Phần B (lntrang)